| 2018.08.20(月) |
(研究成果)コムギのゲノム配列解読を達成 コムギの新品種開発の基盤完成 新品種開発加速化に期待(農研機構) |
|---|
農研機構と京都大学などが参加した国際コンソーシアムは、コムギゲノムの塩基配列解読を達成した。コムギの21本の染色体に相当する配列を構築し、様々な特徴を決定する10万個以上の遺伝子を見出した。この情報を利用し、有用な遺伝子の単離や
DNAマーカー
1)
の開発を通じて、新品種育成が加速すると期待される。
農研機構と国立大学法人京都大学などが参加した国際コムギゲノム解読コンソーシアム (IWGSC) 2) はコムギゲノムの塩基配列の解読に成功。2005年に結成されたIWGSCは、イネやトウモロコシとならぶ世界の三大穀物の一つであるコムギのゲノム解読を目指して活動を進め、2014年には染色体毎に区分された 概要配列 3) を解読し公表した。
しかし、概要配列は細かく分断されており、染色体内での遺伝子位置が不明であったことから、染色体が一本につながった精度の高い 参照ゲノム配列 4) が求められていた。今回、IWGSCはコムギのゲノムの94%をカバーする参照ゲノム配列の解読を達成し、107,891個の遺伝子を見出し、病害抵抗性や小麦粉の品質に関わる遺伝子群の詳細を明らかにした。
コムギは世界中の人たちが日常消費している食物カロリーの約2割を占めており、今回の成果は、人口増加や気候変動による世界的な食糧問題を解決するための作物改良研究の基盤となるとともに、我が国においても縞萎縮病(しまいしゅくびょう)等の各種病害に対する抵抗性品種や製パン性を向上させた高品質品種等の新品種開発の加速化につながる。
研究内容の詳細は、17日(米国時間2018年8月16日午後2時、日本時間2018年8月17日午前3時)国際科学専門誌「Science」電子版に掲載された。関連情報(予算:農林水産省委託プロジェクト「新農業展開ゲノム」および「次世代ゲノム基盤」、運営費交付金)。
このお問い合わせは研究推進責任者:農研機構次世代作物開発研究センター所長 矢野昌裕、研究担当者:農研機構次世代作物開発研究センター基盤研究領域 遺伝子機能解析ユニット長 半田裕一、農研機構次世代作物開発研究センター基盤研究領域 教育種法開発ユニット主任研究員 小林史典、(国)京都大学大学院 農学研究科准教授 那須田周平、広報担当者:農研機構次世代作物開発研究センター広報プランナー 大槻寛まで。
<開発の社会的背景と経緯>
コムギはイネやトウモロコシとともに世界の三大穀物の一つであり、私たち人類が日常消費するカロリーの約2割を担っているとともに、重要なタンパク質源になっている。しかし近年では、地球規模の環境変動や人口増加によって世界のコムギ需給は逼迫しつつあり、今後40年の間に60%以上の増収が必要と言われている。
また、コムギいもち病といった新たな脅威の出現に対応するため、「環境の変動に強く、安定して生産できるコムギ品種」や「収穫量が飛躍的に高いコムギ品種」の開発が急務となっている。また、我が国においても優れた製粉品質を持ち十分な生産量を確保できる良品質高収量の品種や、日本の湿潤な気候等によって多発する穂発芽や赤かび病等に対して十分な耐性を持つ品種の開発が切望されている。
<研究の経緯>
コムギの品種改良を効率的に行うためには、ゲノム情報に基づいて、農業上有用な遺伝子を発見し、形質と連鎖するDNAマーカーを作出することが必要だ。しかし、現在世界で栽培されているコムギ(栽培コムギ)は、異なる3種類の祖先種のゲノムからなる6倍体(AABBDD)と複雑なため解読が難しく、遺伝子構成の全体像がわかっていなかった。
また3種類のゲノムにはそれぞれ、同じ祖先から分岐した塩基配列が似ている 「同祖遺伝子」 5) が存在しているため、ある遺伝子の配列がわかっても、それがAゲノム染色体上にあるのか、あるいはBゲノム染色体やDゲノム染色体にあるのかが分からず、DNAマーカーとしての活用が困難であった。さらにコムギのゲノムサイズは、イネの40倍にも及ぶ大きなもので、倍数体の複雑さと相まって、ゲノム情報利用を難しくしていた。
こうした状況を打開するため、コムギのゲノム情報を染色体毎に高精度に解読することを目的として、2005年に国際コムギゲノム解読コンソーシアム(IWGSC)が組織され、各国の研究者が コムギ品種「チャイニーズスプリング」 6) ( 図1 )の21本(3ゲノム×7本)の染色体を分担して、ゲノム解読を開始した。
日本も農業生物資源研究所(現:農研機構)を中心とした研究グループを組織してIWGSCに参加し、「6B染色体」の解読を担当してきた。IWGSCは2014年にコムギの核から染色体を取り出し、染色体毎に配列の解読を行い、ゲノムの61%に相当する領域をカバーする概要配列を公開した(農業生物資源研究所 平成26年7月17日プレスリリース「コムギのゲノム配列の概要解読に成功」)。しかし概要配列では、ゲノム全体を十分にカバーできていないこと、得られた遺伝子の帰属染色体はわかるものの相互の位置関係が不明であること等から、IWGSCは染色体毎に一本につながった高精度参照ゲノム配列の決定に向けて、引き続き共同研究を進めてきた( 図2 )。
<研究の内容・意義>
IWGSCは、コムギのゲノムの94%をカバーする参照ゲノム配列14.5Gb(145億塩基対)の解読に成功した。この参照ゲノム配列は1.7~8.9kb(1700~8900塩基対)に細かく分断されていた概要配列とは異なり、各染色体について一つながりの配列となっていて(平均6.7億塩基対)、これまで明らかではなかった染色体内での遺伝子の位置関係が明らかになるなど、極めて高精度なものだ。これにより特定領域の中に存在する遺伝子の数、種類や位置が明らかになり、マーカー等で絞り込むことにより、遺伝子単離が容易になるなど、コムギ品種の開発を大きく加速することが可能になる。
例えばこの参照ゲノム配列の解析から、107,891個の遺伝子が推定され、その中には病害抵抗性に関与すると考えられているNB-LRR(nucleotide-binding, leucine rich repeat proteins)遺伝子ファミリーに属するものが2,000個以上、小麦粉の品質に関与する種子貯蔵タンパク質であるプロラミン遺伝子ファミリーに属するものが1,000個以上見出されており、病害抵抗性付与や品質改良への利用が期待される。
また、850におよぶさまざまな部位や生育ステージにおける 「発現遺伝子情報」 7) から、同祖遺伝子の使い分けや協調して発現する遺伝子群が見出され、倍数体における遺伝子機能や遺伝子ネットワーク解明につながる。
<今後の予定・期待>
今回、解読されたコムギの参照ゲノム配列情報を活用することで、「環境の変動に強く、安定して生産できるコムギ品種」や「収穫量が飛躍的に高いコムギ品種」など、国内外でのコムギの新品種の開発が進むことが期待される。
農研機構は、参照ゲノム配列情報を用いて縞萎縮病等の各種病害に対する抵抗性遺伝子や製パン性等の品質を向上させる遺伝子の同定と機能解明を進める。この成果の利用によりDNAマーカー開発が促進され、高品質で安定生産が可能な小麦品種の開発が容易になり、国産小麦の振興に寄与できる。
ゲノム配列やそれに基づくDNAマーカー情報は、農業上有用な遺伝子の特定や、その育種利用だけにとどまらず、これまでにゲノム解読が終了したイネ、トウモロコシ、オオムギ、ソルガム等との比較を通じて、私たちの食糧資源として重要なイネ科作物全体の進化等を理解する糸口を得るなど、基礎的な研究に対しても大きな貢献を果たすものと期待される。
<用語の解説>
1)DNAマーカー
遺伝子の目印となるDNA配列。導入したい形質に関わる遺伝子をDNAマーカーの有無で確認して個体を選抜することが可能となる。さらに従前の形質(例えば「背が高い」「色が濃い」といった見た目の性質)を対象とする選抜とは異なり、環境による影響を受けないので安定して利用できる。
2)国際コムギゲノム解読コンソーシアム(IWGSC; The International Wheat Genome Sequencing Consortium)
国際コムギゲノム解読コンソーシアムは、コムギのゲノム配列解読に興味のある研究者、育種家、栽培農家など、64カ国、610組織、約2100名のメンバーによって構成されている団体で、2005年に結成され、現在はアメリカで法人登録されている。日本はコンソーシアム結成時から加わっており、実際に21本のコムギ染色体のゲノム解読に関わっている16カ国(オーストラリア、カナダ、チェコ、フランス、ドイツ、インド、イスラエル、イタリア、日本、ノルウェー、中国、ロシア、スイス、トルコ、イギリス、アメリカ)と1企業のうちの主要メンバーとして、6B染色体のゲノム解読を担当してきた。
3)概要配列
ゲノム配列中には、解読が困難な部分(例えば繰り返し配列など)等が含まれていたりするために、配列を一つながりにして染色体への対応をつけて全ゲノムの完全な配列を取得するには膨大な労力と時間がかかる。解読率が低く、配列が不連続であったり、正確に配列推定されていない概要配列の状態でも、おおよその遺伝子情報は得ることができるため、さまざまな研究現場で利用されることがある。ドラフ配列とも呼ばれる。
4)参照ゲノム配列
その種のゲノムを代表する塩基配列。この参照配列を基準として、各品種や各個体の塩基配列と比較することにより、品種等に見られる形質の変異をもたらす遺伝子配列の違いを検出することができる。
5)同祖遺伝子
祖先を共通にする遺伝子。コムギは共通の祖先種から分化した3つの2倍体種が、後に交雑して成立した6倍体(AABBDD)( 図3 )。これら3つの2倍体種のゲノム(Aゲノム、Bゲノム、Dゲノム)も、共通祖先種のゲノムから分化してきたため、それぞれのゲノムには共通祖先に由来する遺伝子があり、それを同祖遺伝子と呼ぶ。
6)コムギ品種「チャイニーズスプリング(Chinese Spring)」
コムギの遺伝学において、20世紀初頭から広く使われてきた栽培コムギの実験用品種。交配操作が容易なことから、1920年代から北米を中心に広く遺伝学の材料として使われるようになり、染色体操作系統などさまざまな派生系統がチャイニーズスプリングを基に作られている。チャイニーズという名が付いているが、中国との直接の関係については必ずしも定かではない。
7)発現遺伝子情報
細胞中のゲノムDNAにコードされている遺伝子から、mRNAに転写されている遺伝子に関する情報。ゲノム配列から遺伝子であることは推測できるが、実際に機能を持ち働いている遺伝子であることをより正確に判断するためには、転写されていることを確認する必要がある。ここでは細胞中の転写されたmRNAを抽出し、その相補的DNA(cDNA)を網羅的に次世代シークエンサーで解析した情報(mRNA-Seq)を発現遺伝子の情報として利用している。
<発表論文>
International Wheat Genome Sequencing Consortium. (2018) Shifting the limits in wheat research and breeding through a fully annotated and anchored reference genome sequence. Science doi: 10.1126/science.aar7191
参考図

図1. 栽培コムギ(Triticum aestivum L.)
背景は現在の日本の基幹品種「きたほなみ」、枠内が今回ゲノム解読を終了した「チャイニーズスプリング」だ。
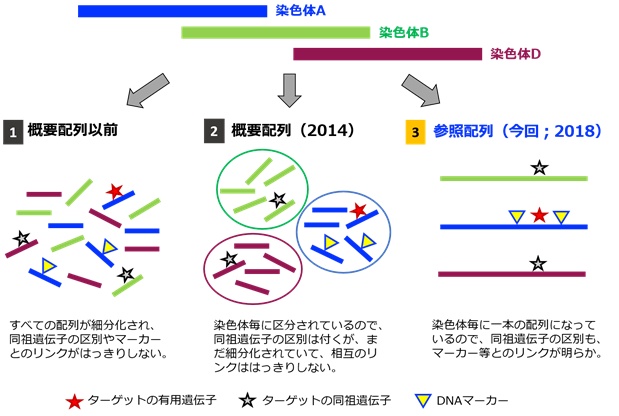
図2. 解読レベルによるコムギゲノム配列情報の違い
共通の祖先から分化してきた三つのゲノムを区別して、さらにそれを一つながりにしていくことで、より正確で使いやすいゲノム情報を得ることができる。

図3. コムギの系統関係
共通の祖先から分化してきた三つの2倍体種(一粒系コムギ、クサビコムギ、タルホコムギ)が相互に交雑してできたのが、現在私たちがパンやうどんとして利用している栽培コムギ(Triticum aestivum L.)である。
この件のお問い合わせは農研機構 広報課まで(電話029-838-8988)。
農研機構と国立大学法人京都大学などが参加した国際コムギゲノム解読コンソーシアム (IWGSC) 2) はコムギゲノムの塩基配列の解読に成功。2005年に結成されたIWGSCは、イネやトウモロコシとならぶ世界の三大穀物の一つであるコムギのゲノム解読を目指して活動を進め、2014年には染色体毎に区分された 概要配列 3) を解読し公表した。
しかし、概要配列は細かく分断されており、染色体内での遺伝子位置が不明であったことから、染色体が一本につながった精度の高い 参照ゲノム配列 4) が求められていた。今回、IWGSCはコムギのゲノムの94%をカバーする参照ゲノム配列の解読を達成し、107,891個の遺伝子を見出し、病害抵抗性や小麦粉の品質に関わる遺伝子群の詳細を明らかにした。
コムギは世界中の人たちが日常消費している食物カロリーの約2割を占めており、今回の成果は、人口増加や気候変動による世界的な食糧問題を解決するための作物改良研究の基盤となるとともに、我が国においても縞萎縮病(しまいしゅくびょう)等の各種病害に対する抵抗性品種や製パン性を向上させた高品質品種等の新品種開発の加速化につながる。
研究内容の詳細は、17日(米国時間2018年8月16日午後2時、日本時間2018年8月17日午前3時)国際科学専門誌「Science」電子版に掲載された。関連情報(予算:農林水産省委託プロジェクト「新農業展開ゲノム」および「次世代ゲノム基盤」、運営費交付金)。
このお問い合わせは研究推進責任者:農研機構次世代作物開発研究センター所長 矢野昌裕、研究担当者:農研機構次世代作物開発研究センター基盤研究領域 遺伝子機能解析ユニット長 半田裕一、農研機構次世代作物開発研究センター基盤研究領域 教育種法開発ユニット主任研究員 小林史典、(国)京都大学大学院 農学研究科准教授 那須田周平、広報担当者:農研機構次世代作物開発研究センター広報プランナー 大槻寛まで。
<開発の社会的背景と経緯>
コムギはイネやトウモロコシとともに世界の三大穀物の一つであり、私たち人類が日常消費するカロリーの約2割を担っているとともに、重要なタンパク質源になっている。しかし近年では、地球規模の環境変動や人口増加によって世界のコムギ需給は逼迫しつつあり、今後40年の間に60%以上の増収が必要と言われている。
また、コムギいもち病といった新たな脅威の出現に対応するため、「環境の変動に強く、安定して生産できるコムギ品種」や「収穫量が飛躍的に高いコムギ品種」の開発が急務となっている。また、我が国においても優れた製粉品質を持ち十分な生産量を確保できる良品質高収量の品種や、日本の湿潤な気候等によって多発する穂発芽や赤かび病等に対して十分な耐性を持つ品種の開発が切望されている。
<研究の経緯>
コムギの品種改良を効率的に行うためには、ゲノム情報に基づいて、農業上有用な遺伝子を発見し、形質と連鎖するDNAマーカーを作出することが必要だ。しかし、現在世界で栽培されているコムギ(栽培コムギ)は、異なる3種類の祖先種のゲノムからなる6倍体(AABBDD)と複雑なため解読が難しく、遺伝子構成の全体像がわかっていなかった。
また3種類のゲノムにはそれぞれ、同じ祖先から分岐した塩基配列が似ている 「同祖遺伝子」 5) が存在しているため、ある遺伝子の配列がわかっても、それがAゲノム染色体上にあるのか、あるいはBゲノム染色体やDゲノム染色体にあるのかが分からず、DNAマーカーとしての活用が困難であった。さらにコムギのゲノムサイズは、イネの40倍にも及ぶ大きなもので、倍数体の複雑さと相まって、ゲノム情報利用を難しくしていた。
こうした状況を打開するため、コムギのゲノム情報を染色体毎に高精度に解読することを目的として、2005年に国際コムギゲノム解読コンソーシアム(IWGSC)が組織され、各国の研究者が コムギ品種「チャイニーズスプリング」 6) ( 図1 )の21本(3ゲノム×7本)の染色体を分担して、ゲノム解読を開始した。
日本も農業生物資源研究所(現:農研機構)を中心とした研究グループを組織してIWGSCに参加し、「6B染色体」の解読を担当してきた。IWGSCは2014年にコムギの核から染色体を取り出し、染色体毎に配列の解読を行い、ゲノムの61%に相当する領域をカバーする概要配列を公開した(農業生物資源研究所 平成26年7月17日プレスリリース「コムギのゲノム配列の概要解読に成功」)。しかし概要配列では、ゲノム全体を十分にカバーできていないこと、得られた遺伝子の帰属染色体はわかるものの相互の位置関係が不明であること等から、IWGSCは染色体毎に一本につながった高精度参照ゲノム配列の決定に向けて、引き続き共同研究を進めてきた( 図2 )。
<研究の内容・意義>
IWGSCは、コムギのゲノムの94%をカバーする参照ゲノム配列14.5Gb(145億塩基対)の解読に成功した。この参照ゲノム配列は1.7~8.9kb(1700~8900塩基対)に細かく分断されていた概要配列とは異なり、各染色体について一つながりの配列となっていて(平均6.7億塩基対)、これまで明らかではなかった染色体内での遺伝子の位置関係が明らかになるなど、極めて高精度なものだ。これにより特定領域の中に存在する遺伝子の数、種類や位置が明らかになり、マーカー等で絞り込むことにより、遺伝子単離が容易になるなど、コムギ品種の開発を大きく加速することが可能になる。
例えばこの参照ゲノム配列の解析から、107,891個の遺伝子が推定され、その中には病害抵抗性に関与すると考えられているNB-LRR(nucleotide-binding, leucine rich repeat proteins)遺伝子ファミリーに属するものが2,000個以上、小麦粉の品質に関与する種子貯蔵タンパク質であるプロラミン遺伝子ファミリーに属するものが1,000個以上見出されており、病害抵抗性付与や品質改良への利用が期待される。
また、850におよぶさまざまな部位や生育ステージにおける 「発現遺伝子情報」 7) から、同祖遺伝子の使い分けや協調して発現する遺伝子群が見出され、倍数体における遺伝子機能や遺伝子ネットワーク解明につながる。
<今後の予定・期待>
今回、解読されたコムギの参照ゲノム配列情報を活用することで、「環境の変動に強く、安定して生産できるコムギ品種」や「収穫量が飛躍的に高いコムギ品種」など、国内外でのコムギの新品種の開発が進むことが期待される。
農研機構は、参照ゲノム配列情報を用いて縞萎縮病等の各種病害に対する抵抗性遺伝子や製パン性等の品質を向上させる遺伝子の同定と機能解明を進める。この成果の利用によりDNAマーカー開発が促進され、高品質で安定生産が可能な小麦品種の開発が容易になり、国産小麦の振興に寄与できる。
ゲノム配列やそれに基づくDNAマーカー情報は、農業上有用な遺伝子の特定や、その育種利用だけにとどまらず、これまでにゲノム解読が終了したイネ、トウモロコシ、オオムギ、ソルガム等との比較を通じて、私たちの食糧資源として重要なイネ科作物全体の進化等を理解する糸口を得るなど、基礎的な研究に対しても大きな貢献を果たすものと期待される。
<用語の解説>
1)DNAマーカー
遺伝子の目印となるDNA配列。導入したい形質に関わる遺伝子をDNAマーカーの有無で確認して個体を選抜することが可能となる。さらに従前の形質(例えば「背が高い」「色が濃い」といった見た目の性質)を対象とする選抜とは異なり、環境による影響を受けないので安定して利用できる。
2)国際コムギゲノム解読コンソーシアム(IWGSC; The International Wheat Genome Sequencing Consortium)
国際コムギゲノム解読コンソーシアムは、コムギのゲノム配列解読に興味のある研究者、育種家、栽培農家など、64カ国、610組織、約2100名のメンバーによって構成されている団体で、2005年に結成され、現在はアメリカで法人登録されている。日本はコンソーシアム結成時から加わっており、実際に21本のコムギ染色体のゲノム解読に関わっている16カ国(オーストラリア、カナダ、チェコ、フランス、ドイツ、インド、イスラエル、イタリア、日本、ノルウェー、中国、ロシア、スイス、トルコ、イギリス、アメリカ)と1企業のうちの主要メンバーとして、6B染色体のゲノム解読を担当してきた。
3)概要配列
ゲノム配列中には、解読が困難な部分(例えば繰り返し配列など)等が含まれていたりするために、配列を一つながりにして染色体への対応をつけて全ゲノムの完全な配列を取得するには膨大な労力と時間がかかる。解読率が低く、配列が不連続であったり、正確に配列推定されていない概要配列の状態でも、おおよその遺伝子情報は得ることができるため、さまざまな研究現場で利用されることがある。ドラフ配列とも呼ばれる。
4)参照ゲノム配列
その種のゲノムを代表する塩基配列。この参照配列を基準として、各品種や各個体の塩基配列と比較することにより、品種等に見られる形質の変異をもたらす遺伝子配列の違いを検出することができる。
5)同祖遺伝子
祖先を共通にする遺伝子。コムギは共通の祖先種から分化した3つの2倍体種が、後に交雑して成立した6倍体(AABBDD)( 図3 )。これら3つの2倍体種のゲノム(Aゲノム、Bゲノム、Dゲノム)も、共通祖先種のゲノムから分化してきたため、それぞれのゲノムには共通祖先に由来する遺伝子があり、それを同祖遺伝子と呼ぶ。
6)コムギ品種「チャイニーズスプリング(Chinese Spring)」
コムギの遺伝学において、20世紀初頭から広く使われてきた栽培コムギの実験用品種。交配操作が容易なことから、1920年代から北米を中心に広く遺伝学の材料として使われるようになり、染色体操作系統などさまざまな派生系統がチャイニーズスプリングを基に作られている。チャイニーズという名が付いているが、中国との直接の関係については必ずしも定かではない。
7)発現遺伝子情報
細胞中のゲノムDNAにコードされている遺伝子から、mRNAに転写されている遺伝子に関する情報。ゲノム配列から遺伝子であることは推測できるが、実際に機能を持ち働いている遺伝子であることをより正確に判断するためには、転写されていることを確認する必要がある。ここでは細胞中の転写されたmRNAを抽出し、その相補的DNA(cDNA)を網羅的に次世代シークエンサーで解析した情報(mRNA-Seq)を発現遺伝子の情報として利用している。
<発表論文>
International Wheat Genome Sequencing Consortium. (2018) Shifting the limits in wheat research and breeding through a fully annotated and anchored reference genome sequence. Science doi: 10.1126/science.aar7191
参考図

図1. 栽培コムギ(Triticum aestivum L.)
背景は現在の日本の基幹品種「きたほなみ」、枠内が今回ゲノム解読を終了した「チャイニーズスプリング」だ。

図2. 解読レベルによるコムギゲノム配列情報の違い
共通の祖先から分化してきた三つのゲノムを区別して、さらにそれを一つながりにしていくことで、より正確で使いやすいゲノム情報を得ることができる。

図3. コムギの系統関係
共通の祖先から分化してきた三つの2倍体種(一粒系コムギ、クサビコムギ、タルホコムギ)が相互に交雑してできたのが、現在私たちがパンやうどんとして利用している栽培コムギ(Triticum aestivum L.)である。
この件のお問い合わせは農研機構 広報課まで(電話029-838-8988)。